
Fylogeni fortolkning, typer af træer, applikationer
EN fylogeni, I evolutionærbiologi er det en repræsentation af den evolutionære historie for en gruppe organismer eller en art, der understreger nedstigningslinjen og slægtskabsforholdet mellem grupperne..
I dag har biologer primært brugt data fra komparativ morfologi og anatomi og fra gensekvenser til at rekonstruere tusinder og tusinder af træer..
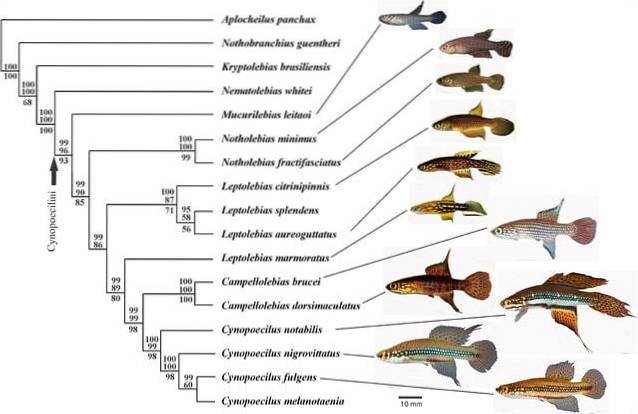
Disse træer søger at beskrive udviklingshistorien for de forskellige dyrearter, planter, mikrober og andre organiske væsener, der bebor jorden..
Analogien med livets træ stammer fra Charles Darwins tid. Denne strålende britiske naturforsker indfanger mesterværket "Arternes oprindelse"Et enkelt billede: et" træ ", der repræsenterer forgreningen af slægterne, startende fra en fælles forfader.
Artikelindeks
- 1 Hvad er fylogeni?
- 2 Hvad er et fylogenetisk træ?
- 3 Hvordan fortolkes fylogenetiske træer?
- 4 Hvordan rekonstrueres fylogenier?
- 4.1 Homologe tegn
- 5 Typer af træer
- 6 Politomias
- 7 Evolutionær klassificering
- 7.1 Monofyletiske slægter
- 7.2 Parafyletiske og polyfyletiske slægter
- 8 applikationer
- 9 Referencer
Hvad er en fylogeni?
I lyset af de biologiske videnskaber er en af de mest fantastiske begivenheder, der har fundet sted, evolution. Denne ændring i organiske former over tid kan repræsenteres i et fylogenetisk træ. Derfor udtrykker fylogeni slægternes historie og hvordan de har ændret sig over tid..
En af de direkte implikationer af denne graf er fælles herkomst. Det vil sige, at alle de organismer, som vi ser i dag, er opstået som efterkommere med ændringer af tidligere former. Denne idé har været en af de mest betydningsfulde i videnskabens historie.
Alle livsformer, som vi kan sætte pris på i dag - fra mikroskopiske bakterier til planter og de største hvirveldyr - er forbundet, og dette forhold er repræsenteret i det store og indviklede livets træ..
Inden for analogien af træet ville de arter, der lever i dag, repræsentere bladene og resten af grenene være deres evolutionære historie.
Hvad er et fylogenetisk træ?
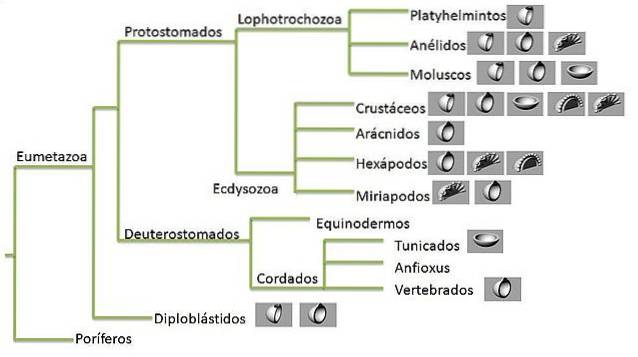
Et fylogenetisk træ er en grafisk repræsentation af en gruppe organismeres evolutionære historie. Dette mønster af historiske forhold er fylogenien, som forskere forsøger at estimere..
Træer består af noder, der forbinder "grene". Terminalknudepunkterne for hver gren er terminal taxa og repræsenterer de sekvenser eller organismer, som data er kendt for - disse kan være levende eller uddøde arter.
De interne knudepunkter repræsenterer hypotetiske forfædre, mens den forfader, der findes ved roden af træet, repræsenterer forfædren til alle de sekvenser, der er repræsenteret i grafen..
Hvordan fortolkes fylogenetiske træer?
Der er mange måder at repræsentere et fylogenetisk træ på. Derfor er det vigtigt at vide, hvordan man genkender, om disse forskelle, der observeres mellem to træer, skyldes forskellige topologier - det vil sige reelle forskelle svarende til to stavemåder - eller simpelthen er forskelle relateret til repræsentationsstil..
For eksempel kan rækkefølgen, hvor etiketterne vises øverst, variere uden at ændre betydningen af den grafiske repræsentation, generelt navnet på arten, slægten, familien og andre kategorier..
Dette sker, fordi træerne ligner en mobil, hvor grenene kan rotere uden at ændre forholdet mellem de repræsenterede arter..
I denne forstand betyder det ikke noget, hvor mange gange rækkefølgen ændres, eller objekterne, der "hænger", roteres, da det ikke ændrer den måde, de er forbundet på - og det er det vigtige..
Hvordan rekonstrueres fylogenier?
Fylogenier er hypoteser, der er formuleret baseret på indirekte beviser. At belyse en fylogeni ligner en efterforskers job at løse en forbrydelse ved at følge sporene fra gerningsstedet.
Biologer postulerer ofte deres fylogenier ved hjælp af viden fra forskellige grene, såsom paleontologi, komparativ anatomi, komparativ embryologi og molekylærbiologi..
Den fossile optegnelse, selvom den er ufuldstændig, giver meget værdifuld information om divergens tiderne for grupper af arter.
Med tiden har molekylærbiologi overgået alle de førnævnte felter, og de fleste fylogenier udledes af molekylære data..
Målet med at rekonstruere et fylogenetisk træ har en række store ulemper. Der er cirka 1,8 millioner navngivne arter og mange flere uden at blive beskrevet.
Og selvom et betydeligt antal forskere hver dag stræber efter at rekonstruere forholdet mellem arter, er der stadig ikke noget komplet træ.
Homologe karakterer
Når biologer ønsker at beskrive lighederne mellem to strukturer eller processer, kan de gøre det med hensyn til fælles herkomst (homologier), analogier (funktion) eller homoplasi (morfologisk lighed)..
For at rekonstruere en fylogeni anvendes udelukkende homologe tegn. Homologi er et nøglebegreb i evolutionen og i genskabelsen af forholdet mellem arter, da kun det i tilstrækkelig grad afspejler organismernes fælles herkomst.
Antag, at vi ønsker at udlede fylogenien fra tre grupper: fugle, flagermus og mennesker. For at opfylde vores mål besluttede vi at bruge de øvre ekstremiteter som et kendetegn, der hjælper os med at skelne mønsteret af relationer..
Da fugle og flagermus har ændret strukturer til flyvning, kunne vi fejlagtigt konkludere, at flagermus og fugle er tættere forbundet med hinanden end flagermus til mennesker. Hvorfor er vi kommet til den forkerte konklusion? Fordi vi har brugt en analog og ikke-homolog karakter.
For at finde det rigtige forhold skal jeg lede efter en homolog karakter, såsom tilstedeværelsen af hår, brystkirtler og tre små knogler i mellemøret - bare for at nævne nogle få. Imidlertid er homologier ikke lette at diagnosticere.
Typer af træer
Ikke alle træer er ens, der er forskellige grafiske fremstillinger, og hver enkelt formår at inkorporere nogle ejendommelige karakteristika ved gruppens udvikling.
De mest basale træer er cladogrammer. Disse grafer viser forholdet i form af fælles forfædre (ifølge de seneste fælles forfædre).
Tilsætnings træer indeholder yderligere oplysninger og er repræsenteret i grenernes længde.
Tallene, der er knyttet til hver gren, svarer til nogle attributter i sekvensen - såsom mængden af evolutionær ændring, som organismer har gennemgået. Ud over "additive træer" er de også kendt som metriske træer eller fylogrammer..
Ultrametriske træer, også kaldet dendogrammer, er et særligt tilfælde af additivtræer, hvor spidserne på træet er lige langt fra roden til træet.
Disse sidste to varianter har alle de data, vi kan finde i et cladogram, og ekstra information. Derfor er de ikke eksklusive, hvis ikke komplementære.
Politomias
Mange gange løses ikke knuderne på træerne fuldt ud. Visuelt siges det, at der er en polytomi, når mere end tre grene dukker op fra en ny (der er en enkelt forfader til mere end to umiddelbare efterkommere). Når et træ ikke har polytomier, siges det at være helt løst.
Der er to typer polytomier. Den første er de "hårde" polytomier. Disse er iboende for studiegruppen og indikerer, at efterkommerne udviklede sig på samme tid. Alternativt indikerer "bløde" polytomier uløste forhold forårsaget af data i sig selv.
Evolutionær klassificering
Monofyletiske slægter
Evolutionære biologer søger at finde en klassifikation, der passer til forgreningsmønsteret for gruppers fylogenetiske historie. I denne proces er der udviklet en række udtryk, der er meget anvendt i evolutionær biologi: monofyletisk, parafyletisk og polyfyletisk..
En monofyletisk taxon eller afstamning er en, der omfatter en forfædres art, som er repræsenteret i knuden, og alle dens efterkommere, men ikke andre arter. Denne gruppering kaldes en klade.
Monofyletiske slægter er defineret på hvert niveau af det taksonomiske hierarki. For eksempel betragtes Family Felidae, en slægt, der indeholder katte (inklusive huskatte), monofyletisk..
Tilsvarende er Animalia også en monofyletisk taxon. Som vi ser familien Felidae er inden for Animalia, så monofyletiske grupper kan indlejres.
Parafyletiske og polyfyletiske slægter
Imidlertid deler ikke alle biologer kladistisk klassifikationstænkning. I tilfælde, hvor dataene ikke er komplette eller blot for nemheds skyld, navngives visse taxaer, der inkluderer arter fra forskellige klader eller højere taxaer, der ikke deler en nyere fælles forfader..
På denne måde er en taxon polyfyletisk, den defineres som en gruppe, der inkluderer organismer fra forskellige klader, og disse deler ikke en fælles forfader. For eksempel, hvis vi ønsker at udpege en gruppe homeotherms, vil det omfatte fugle og pattedyr..
I modsætning hertil indeholder en parafyletisk gruppe ikke alle efterkommere af den seneste fælles forfader. Med andre ord udelukker det nogle af gruppens medlemmer. Det mest anvendte eksempel er krybdyr, denne gruppe indeholder ikke alle efterkommere af den seneste fælles forfader: fugle.
Ansøgninger
Ud over at bidrage til den hårde opgave at belyse livets træ har fylogenier også nogle ret betydningsfulde anvendelser.
På det medicinske område anvendes fylogenier til at spore oprindelses- og overførselshastighederne for infektiøse sygdomme, såsom AIDS, denguefeber og influenza..
De bruges også inden for bevaringsbiologi. Kendskab til fylogenien hos en truet art er vigtig for at spore krydsningsmønstre og niveauet for hybridisering og indavl mellem individer..
Referencer
- Baum, D. A., Smith, S. D., og Donovan, S. S. (2005). Træ-tænkning udfordring. Videnskab, 310(5750), 979-980.
- Curtis, H., & Barnes, N. S. (1994). Invitation til biologi. Macmillan.
- Hall, B. K. (red.). (2012). Homologi: Det hierarkiske grundlag for komparativ biologi. Akademisk presse.
- Hickman, C. P., Roberts, L. S., Larson, A., Ober, W. C., & Garrison, C. (2001). Integrerede zoologiske principper. McGraw-Hill.
- Hinchliff, CE, Smith, SA, Allman, JF, Burleigh, JG, Chaudhary, R., Coghill, LM, Crandall, KA, Deng, J., Drew, BT, Gazis, R., Gude, K., Hibbett, DS, Katz, LA, Laughinghouse, HD, McTavish, EJ, Midford, PE, Owen, CL, Ree, RH, Rees, JA, Soltis, DE, Williams, T.,… Cranston, KA (2015). Syntese af fylogeni og taksonomi til et omfattende livets træ. Forhandlinger fra National Academy of Sciences i Amerikas Forenede Stater, 112(41), 12764-9.
- Kardong, K. V. (2006). Vertebrater: komparativ anatomi, funktion, evolution. McGraw-Hill.
- Page, R. D., & Holmes, E. C. (2009). Molekylær udvikling: en fylogenetisk tilgang. John Wiley & Sons.



Endnu ingen kommentarer